HBsAgGi
M-HBsのO型糖鎖修飾PreS2ドメインに対するモノクローナル抗体で、感染性HBV粒子(DNAビリオンおよびRNAビリオン)を主に認識します。
- 商品コード:RCA-001
- 包装/規格: 50μg/tube
- 該当する遺伝子型: Genotype C
- 希望販売価格: 70,000円(税抜)
HBsAgGi ELISAキット
M-HBsのO型糖鎖修飾PreS2ドメインに対するモノクローナル抗体を用いたELISAキットで、感染性HBV粒子(DNAビリオンおよびRNAビリオン)を測定することが可能です。
- 商品コード:RCEK-001
- 包装/規格: 96 Test
- 該当する遺伝子型: Genotype C
- 希望販売価格: 100,000円(税抜)
HBsAgGiの特長:
HBsAgGiは、日本で最も多い遺伝子型であるgenotype CのHBV感染性粒子を効率的に認識する抗体です。
genotype CのHBV感染性粒子は、M-HBsAg上のPreS2ドメインに特徴的なO型糖鎖修飾がされることが知られており、本抗体はO型糖鎖修飾されたPreS2ドメインを認識します。
既存のHBsAg抗体と異なり、HBVの非感染性粒子を検出しないため、様々な研究に有用です。
- 様々なステージの慢性B 型肝炎患者から採取した検体を、市販のマーカー(HBV-DNA、HBV-RNA、qHBsAg、HBcrAg)と比較することが可能です。HBsAgGi抗体を免疫沈降に用いるとHBV感染性粒子(DNAビリオンおよびRNAビリオン)の濃縮が可能です。(免疫沈降後にHBV-DNAやHBV-RNAの測定をすることで定量も可能です)
- 培養上清から前臨床試験サンプルまで、幅広くHBVを含むサンプルの分析が可能です。PreS2ドメインを認識する抗体ですので、Sドメインの変異によって従来のHBsAg抗体では検出されなくなったHBVの検出にも使用が可能です。
参考文献
| No. | 文献名 | コメント |
|---|---|---|
| 1 | Yao B, et al. Antiviral Res. Jan 7, 2025 | 核酸アナログ治療後のHBV DNAが検出されない患者において、HBsAgGiがHBV RNAと有意に相関することが報告されました。DNA陰性患者において、HBsAgGiがHBV RNAを含む感染性粒子のマーカーとなることが示唆されました。 |
| 2 | Okumura T et al, Hepatol Res. Aug 20, 2024. | 新規バイオマーカーとしてのHBsAgGiの可能性が、Editorialで取り上げられました。 |
| 3 | Yuji I, et al. Hepatol Res. May 28, 2024 | HBV感染者232症例を対象に、HBsAgGi値が将来のHBs抗原消失の予測因子であることが報告されました。 |
| 4 | Ivana L et al. Pathogens 2024 Jan 3;13(1):46 | HBsAgGiが、HBV-RNAやcccDNAのサロゲートマーカーとして使える可能性がReviewで取り上げられました。 |
| 5 | Kozuka R, al. Hepatol Res. Feb 7, 2024. | HBV感染者112症例を対象に縦断的解析を実施、HBsAgGiとqHBsAgの比率が肝癌発症の予測因子であることが報告されました。 |
| 6 | Okumura T, et al. J Viral Hepat. 30: 731-739, 2023 | 通院中のHBV感染者124症例を対象にHBsAgGiを横断的に解析した試験。一部縦断的解析を実施し、癌発症との関連を示唆した初の仕事が報告されました。 |
| 7 | Murata A, et al. BMC Gastroenterol. 22: 270, 2022. | HBsAgGiに対するモノクローナル抗体を活用したELISAを作成(研究試薬としての「HBsAgGi ELISAキット」)し、未治療の慢性B型肝炎47症例を対象とした、核酸アナログ治療前後の血中HBsAgGi値を世界で初めて測定、発表したエポック・メイキング的な論文。ここで既にRNA粒子をキャプチャーすることも報告されました。 |
| 8 | Angata K, et al. Biochim Biophys Acta Gen Subj. 1866:130020, 2022. | HBsAgGiに対するモノクローナル抗体の作成および特性・活性が報告されました。 |
| 9 | Wagatsuma T, et al. Anal Chem. 90:10196-10203. 2018. | HBV感染性粒子に好んで発現する糖鎖構造(M-HBsのPreS2領域のO型糖鎖=HBsAgGi)が特定され、報告されました。 |
HBsAgGi ELISAキット:
M-HBsのO型糖鎖修飾PreS2ドメインに対するモノクローナル抗体を用いたELISAキットで、感染性HBV粒子(DNAビリオンおよびRNAビリオン)を測定することが可能です。
* 当社より直接ご購入いただけます。
該当する遺伝子型: C
96 Test
商品コード: RCEK-001
希望販売価格: 100,000円 (税抜)
概要
HBsAg糖鎖異性体に対する組換えモノクローナル抗体(HBsAgGi)は、主に感染性HBV粒子に存在するM-HBs上のO型糖鎖修飾PreS2ドメインを特異的に認識します。非感染性粒子を含む、すべてのHBV粒子を認識する従来のHBsAg検査と比較して、HBsAgGiは、感染性粒子(DNAビリオンおよびRNAビリオン)のみを認識します。
当社では、HBsAgGiを用いたHBV感染性粒子を測定する新しいELISAシステムを初めて確立しました。HBV患者の血清には、感染性HBV粒子よりも非感染性粒子が多く含まれていますが、感染性粒子と非感染性粒子の比率は患者ごとに異なります。従って、感染性HBV粒子の測定は、新たな患者の病態解析に有効です。
ELISAキットの構成
HBsAgGi antibody coated plate:96 well (8-well strip x 12)
Standard M-HBsAg
Dilution Buffer
HRP-labeled HBsAgGi antibody
20X Wash Buffer
TMB Substrate
Stop Solution
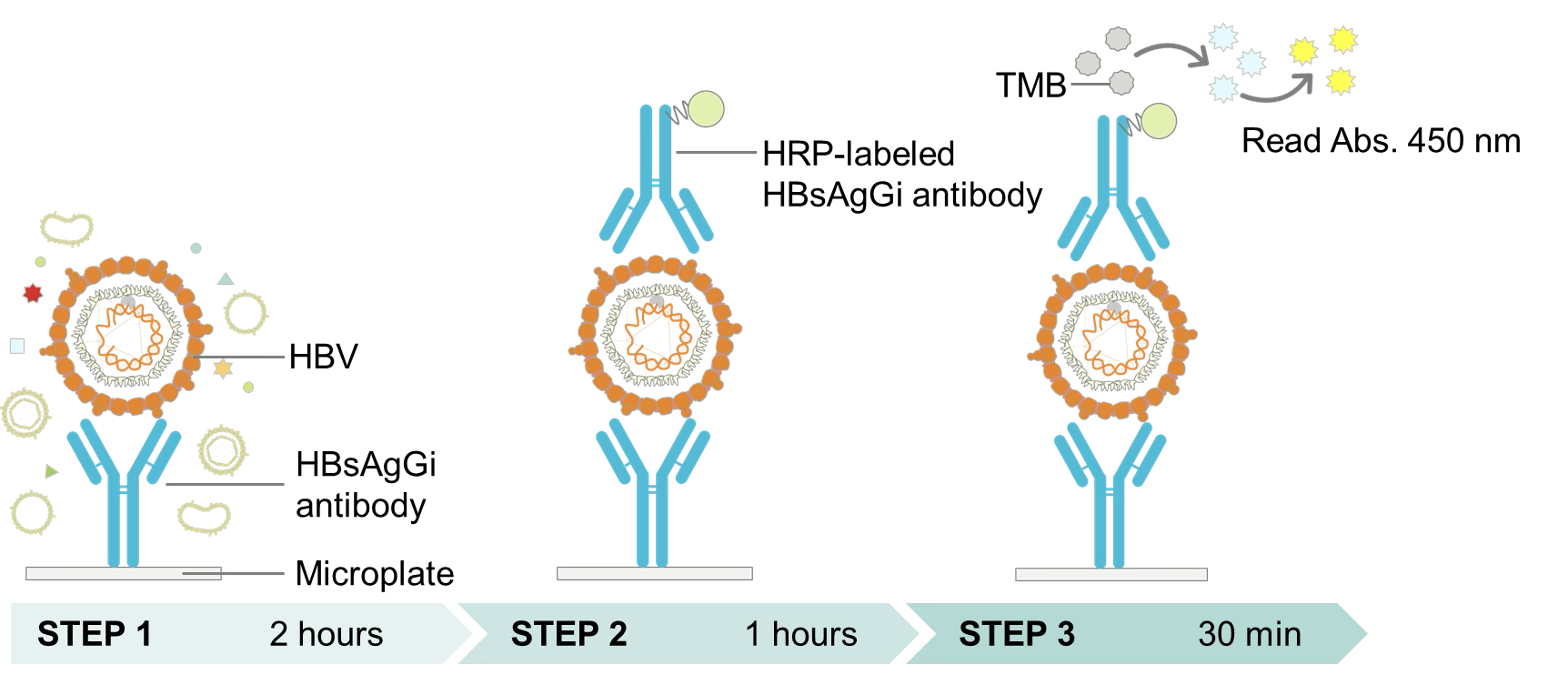
HBsAgGi ELISAキットによるHBV DNA粒子の測定は3つのステップで行います
HBV感染性粒子は先ずHBsAgGi抗体によって捕捉されます(STEP1)。洗浄後、ELISA用マイクロウェルに捕捉されたHBV感染性粒子は、さらにHRP標識HBsAgGi抗体と反応します(STEP2)。さらに洗浄後、HRP基質(TMB基質)をウェルに添加します。インキュベーション後、反応を停止し、プレートリーダーで450nmの吸光度を測定します(STEP 3)。
Safety data sheet (SDS) / ユーザーガイド
参考文献:
1. Schadler and Hildt (2009) Viruses 1:185-209.
2. Schmitt et al. (2004) J Gen Virol 85:2045-2053.
3. Dobrica et al. (2020) Cells 9:1404.
4. Wagatsuma et al. (2018) Anal Chem 90:10196-10203.
5. Angata et al. (2021) Biochim Biophys Acta Gen Subj. 1866:130020
HBsAgGi:
HBs抗原の糖鎖異性体

M-HBsのO型糖鎖修飾PreS2ドメインに対するモノクローナル抗体で、感染性HBV粒子(DNAビリオンおよびRNAビリオン)を主に認識します。
* 当社より直接ご購入いただけます。
該当する遺伝子型: C
50μg
商品コード: RCA-001
希望販売価格: 70,000円(税抜)
概要
HBVエンベロープタンパク質は、PreS1、PreS2、Sドメインを含むHBsAg遺伝子から作られるS-、M-、L-HBsの3種類の表面抗原で構成されています1。 HBsAgはN型糖鎖やO型糖鎖で修飾されています2,3。HBsAgの全糖鎖構造解析の結果、M-HBsのPreS2ドメインはO型糖鎖で修飾されており、L-HBsのPreS2ドメインにはほとんど存在しないこと、O型糖鎖の修飾部位は遺伝子型C(gC)に高度に保存されていることが明らかになりました4,5.
HBsAgの糖鎖異性体に対する組換えモノクローナル抗体(HBsAgGi)は、O型糖鎖を持つPreS2糖ペプチドを用いて作製されました。HBsAgGiは、すべてのHBV粒子を認識する従来のHBsAg検査と比較して、感染性HBV粒子(DNAビリオンおよびRNAビリオン)を特異的に認識します。
HBsAgGi構成
⚫︎ アイソタイプ:マウスIgG1
⚫︎ 軽鎖: κ(カッパ)
⚫︎ 用量: 50μg
⚫︎ 保存方法: 短期間は4℃で保存してください。長期保存の場合は-20℃で保存し、凍結融解は避けてください。
HBsAgGiの用途
ウエスタン
HBV粒子の免疫沈降・分離
ELISA法
HBV感染阻害アッセイ
免疫組織化学
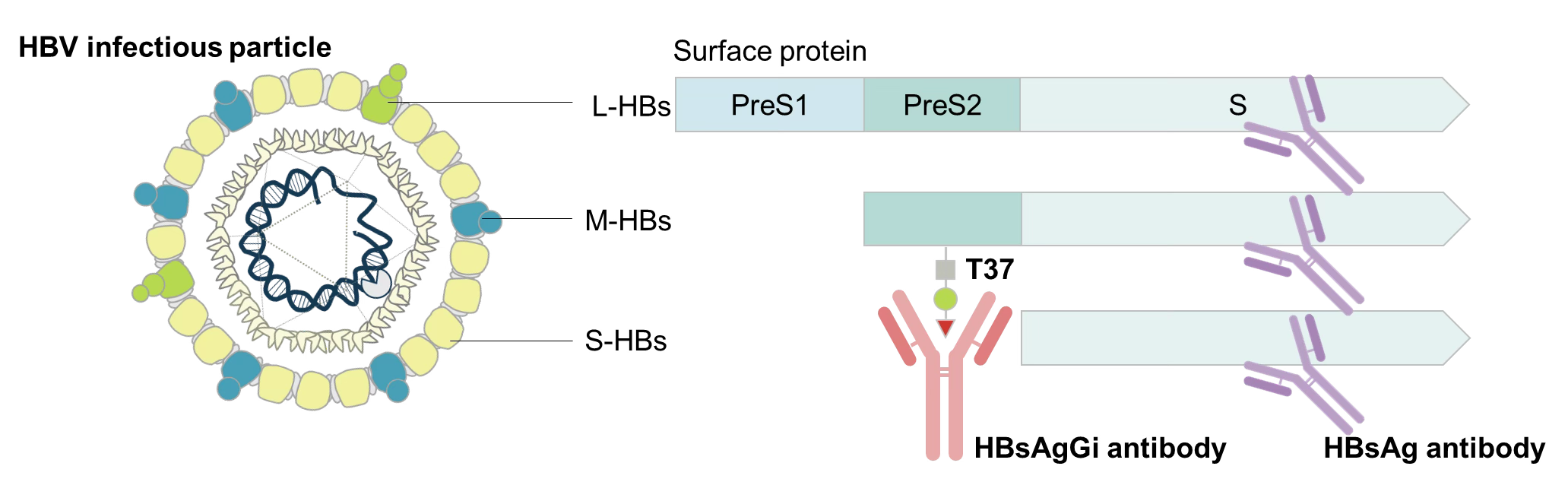
HBsAgGiは、HBV感染性粒子中のO型糖鎖修飾されたM-HBsAgを特異的に認識します
HBVは、エンベロープタンパク質(HBsAg)、HBVコア、HBV DNAから構成されています。HBsAg遺伝子は、PreS1、PreS2、S-ドメインから構成されています。
HBsAg糖鎖異性体に対する組換えモノクローナル抗体(HBsAgGi)は、主に感染性HBV粒子に存在するM-HBs上のO型糖鎖修飾PreS2ドメインを特異的に認識します。
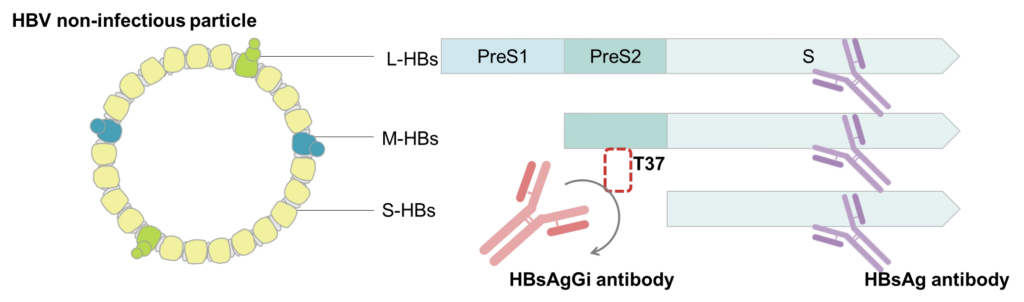
HBV患者の血清中には、感染性粒子(DNAビリオンおよびRNAビリオン)よりも非感染性粒子の方がはるかに多く存在します。既存の抗HBsAg抗体が非感染性粒子を含む全てのHBV粒子を認識するのに対し、HBsAgGiは感染性HBV粒子を認識するため、HBsAgGiの測定により新たな研究が実施可能です。
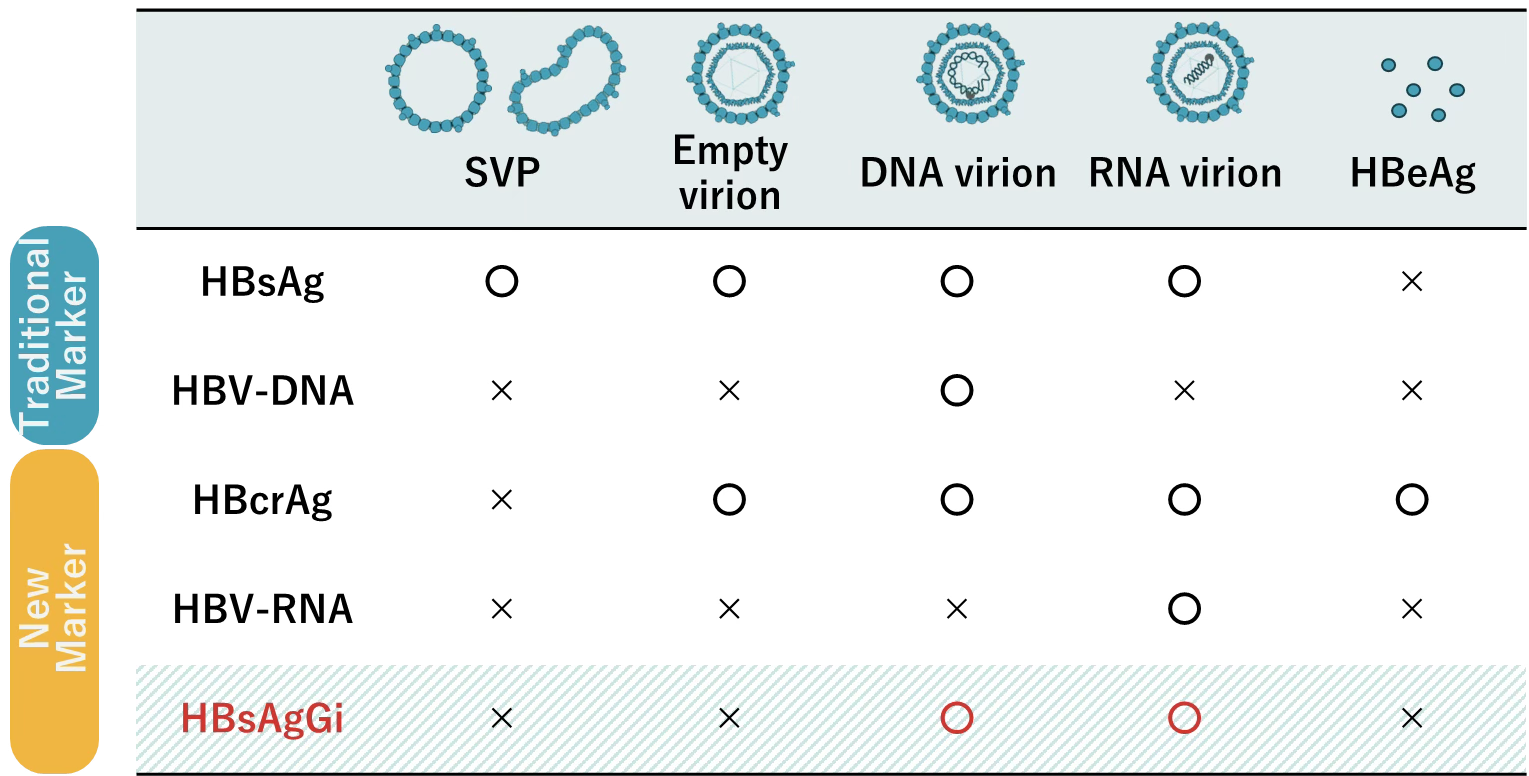
HBsAgGiとその他の感染性HBVを検出するためのマーカー
HBVマーカーの比較表。従来、HBV患者の状態の把握のためにはqHBsAgやHBV DNAの測定が行われてきましたが、近年ではHBcrAgやHBV RNAが患者の状態を診断する新しいマーカーとして注目されつつあります。対象となるHBVの粒子や分子は表のとおりです。〇印はターゲットとなる粒子・分子、×印はそのマーカーが認識できない、あるいは推奨できないことを表しています。
HBsAgGiは、M-HBsAgを含む粒子、例えばDNAやRNAを含むHBV粒子を検出する事ができます。
参考文献:
1. Schadler and Hildt (2009) Viruses 1:185-209.
2. Schmitt et al. (2004) J Gen Virol 85:2045-2053.
3. Dobrica et al. (2020) Cells 9:1404.
4. Wagatsuma et al. (2018) Anal Chem 90:10196-10203.
5. Angata et al. (2021) Biochim Biophys Acta Gen Subj. 1866:130020


